Enfoque Genético de las Enfermedades Renales Hereditarias
Palabras clave
Diagnóstico genético
1. INTRODUCCIÓN
2. GENOMA HUMANO Y SUS VARIANTES DE SECUENCIA
3. ENFERMEDADES GENÉTICAS Y PATRONES DE HERENCIA
4. MÉTODOS DE DIAGNÓSTICO GENÉTICO
5. NOMENCLATURA Y CLASIFICACIÓN DE VARIANTES DE SECUENCIA
6. FACTORES QUE CONTRIBUYEN A LA VARIABILIDAD FENOTÍPICA DE LAS ERH
7. UTILIDADES CLÍNICAS DEL DIAGNÓSTICO GENÉTICO EN NEFROLOGÍA
8. BIBLIOGRAFÍA
9. WEBGRAFÍA
1. INTRODUCCIÓN
Las enfermedades genéticas representan aproximadamente el 80% de las enfermedades raras o minoritarias, definidas como aquellas que tienen una prevalencia menor de 1 en 2000 personas. Se estima que hay entre 6.000 y 8.000 enfermedades raras, con 250-280 nuevas enfermedades descritas anualmente, que afectan aproximadamente al 6-8% de la población. La rareza de estas enfermedades individualmente implica desafíos significativos para los pacientes afectados, sus familias y para sus médicos que intentan conseguir un diagnóstico etiológico definitivo. Las enfermedades raras mayoritariamente se diagnostican en la edad pediátrica y son responsables del 35% de muertes en el primer año de vida. Se carece de tratamientos específicos efectivos para la gran mayoría de estas enfermedades, y cuando existen, a menudo son muy caros debido al esfuerzo científico que implica desarrollarlos, y al pequeño potencial de mercado que tienen [1].
Las enfermedades renales raras comprenden más de 150 enfermedades diferentes, tienen una prevalencia global de alrededor de 60 a 80 casos por cada 100.000 individuos en Europa y EE.UU. y, mayoritariamente son hereditarias. Individualmente todas las enfermedades renales hereditarias (ERH) son enfermedades raras, excepto la poliquistosis renal autosómica dominante (PQRAD). Globalmente, las ERH son la quinta causa más común de fallo renal después de la diabetes, hipertensión, glomerulonefritis y pielonefritis. Al menos el 10% de los adultos y la mayoría de niños que requieren terapia renal sustitutiva (TRS) tienen una ERH [2] [3].
El desarrollo e implantación de las técnicas de análisis genómico que ha sucedido en las dos últimas décadas, ha permitido el descubrimiento de un gran número de genes causantes de ERH. Actualmente, se conocen más de 600 genes causantes o asociados a ERH [4] [5]. Este avance en el conocimiento genético de las ERH está ofreciendo nuevas herramientas para su diagnóstico y manejo, así como cambios en su nomenclatura y clasificación. Algunas ERH están siendo renombradas con una nueva terminología que incluye el nombre del gen causante, como la nefropatía HNF1B [6], o la nefropatía túbulo-intersticial autosómica dominante (NTAD) causada por el gen UMOD (NTAD-UMOD), MUC1 (NTAD-MUC1), REN (NTAD-REN), HNF1B (NTAD-HNF1B), SEC61A1 (NTAD-SEC61A1) [7], y que clásicamente se denominaba Enfermedad Quística Medular.
El diagnóstico clínico de las ERH puede ser complicado debido a su elevada heterogeneidad clínica (edad de inicio, gravedad y progresión de los síntomas, manifestaciones extrarrenales) y al considerable solapamiento de las manifestaciones clínicas entre varias ERH. Por ejemplo, una paciente joven con quistes renales bilaterales sin antecedentes familiares podría estar afectada de PQRAD, de nefropatía HNF1B o incluso de una forma monosintomática de síndrome oro-facio-digital. Existen varios ejemplos de genes inicialmente descritos como causantes de un síndrome que a posteriori se ha identificado que pueden causar únicamente uno de los síntomas. Por ejemplo, el gen LMX1B se describió inicialmente como causante del síndrome de uña-rótula (OMIM #161200, Orphanet #2614), pero a posteriori, se han descrito pacientes con variantes patogénicas en este gen que presentan distintos grados de proteinuria, algunos con biopsia de glomeruloesclerosis segmentaria y focal, y sin alteraciones extrarrenales (OMIM #256020, Orphanet #2613) [8].
Por otro lado, mutaciones en un mismo gen puede causar distintas entidades clínicas. Por ejemplo, el gen PAX2 inicialmente descrito como causante de anomalías congénitas del riñón y el tracto urinario (CAKUT) frecuentemente asociadas a coloboma del nervio óptico, entidad conocida como el síndrome renal-coloboma [9], y a posteriori, también se ha identificado como causante de glomeruloesclerosis segmentaria y focal con patrón autosómico dominante [10].
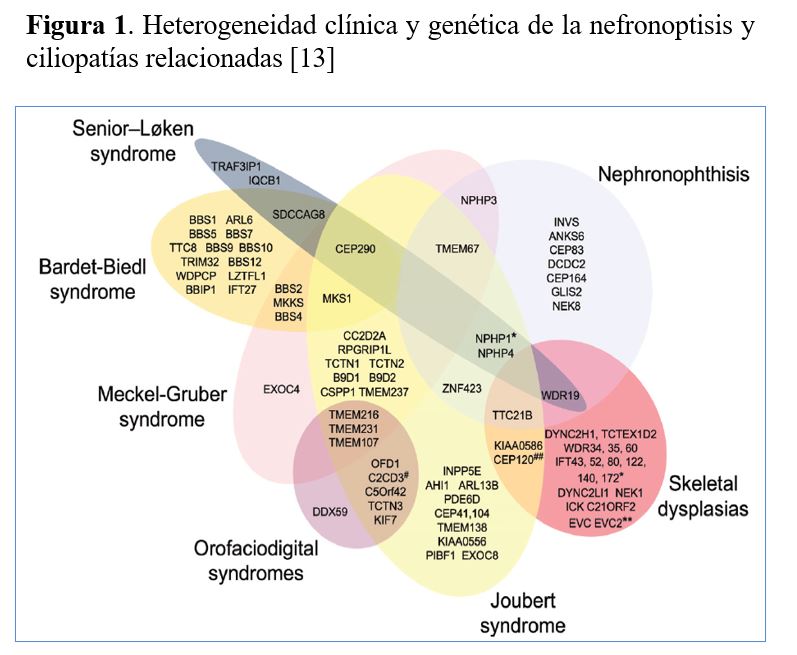
La mayoría de ERH presentan heterogeneidad genética (varios genes causan una misma ERH) que puede ser muy elevada como en la nefronoptisis y ciliopatías relacionadas con más de 20 genes causantes [11] (Figura 1) y el síndrome nefrótico córtico-resistente con más de 50 genes [12].
El diagnóstio genético de una ERH permite un diagnóstico etiológico definitivo, identificando su causa molecular (genotipo), y nos da acceso a importante información en la literatura y en portales de información de referencia de enfermedades genéticas como Online Mendelian Inheritance in Man (OMIM), Orphanet y GeneReviews. Esta información resulta de gran ayuda en enfermedades tan infrecuentes y nos orienta en las manifestaciones clínicas que puede presentar el paciente, en su manejo y opciones terapéuticas, siendo la base de la medicina predictiva y personalizada. Otro punto esencial del diagnóstico genético es que nos permite ofrecer un asesoramiento genético preciso tanto del paciente como de sus familiares.
2. GENOMA HUMANO Y SUS VARIANTES DE SECUENCIA
La secuencia de DNA del genoma humano está contenida en 23 pares de cromosomas en el núcleo de cada célula diploide y contiene la información genética necesaria para el desarrollo y funcionamiento de un ser humano. El genoma humano está formado por 3 billones de pares de bases o nucleótidos [Adenina (A), Citosina (C), Timina (T), Guanina (G)] que contienen unos 20.000 genes. La parte codificante de los genes se denominan exones y al conjunto de todos los exones (~180.000) de todos los genes del genoma se le denomina exoma.
Existe una secuencia de referencia del genoma humano generada por el Proyecto Genoma Humano. La secuencia de referencia fue ensamblada por científicos a partir de la secuenciación de DNA de varios donantes individuales y es de gran utilidad para poder generar comparaciones entre secuencias.
Si bien dos seres humanos del mismo sexo comparten el 99.9% de su secuencia de DNA, aproximadamente cada 1000 nucleótidos existe una variante de secuencia. Cada individuo posee una secuencia única de su genoma, presente en todas las células del cuerpo, fruto de pequeñas variaciones en la combinación de estos 4 nucleótidos, que se han producido y transmitido al largo de las generaciones. Estas variantes fundamentan buena parte de la variabilidad fenotípica interindividual y hacen que no existan dos individuos idénticos, a excepción de los gemelos univitelinos.
La mayoría de estas variantes no alteran la secuencia que codifica para proteína o su nivel de expresión, es decir, que la mayoría son silenciosas y carecen de expresión fenotípica o bien dan lugar a diferencias en el fenotipo que no son patológicas. Estas variantes se denominan variantes benignas (neutras) o clásicamente polimorfismos y generalmente están presentes en una frecuencia superior al 1% en la población general.
En cambio, se denominan variantes patogénicas, clásicamente denominadas mutaciones, a las variantes que alteran la secuencia que codifica para proteína o su nivel de expresión, dando lugar a una falta o ganancia de función proteica, que causan una enfermedad genética. Estas variantes patogénicas se encuentran en una frecuencia muy baja en la población general. Las variantes patogénicas pueden ser cromosómicas, si afectan al número o estructura de los cromosomas, o génicas o genéticas, si alteran un solo gen.
Un alelo es cada una de las formas alternativas que puede tener un mismo gen. Un individuo hereda dos alelos para cada gen autosómico, uno del padre y el otro de la madre. Los alelos se encuentran en la misma posición dentro de los cromosomas homólogos. Se puede distinguir entre los llamados alelos normales o mutados, si han sufrido alguna alteración que hace que su secuencia haya cambiado. Para una determinada variante de secuencia un individuo puede ser: homocigoto, si tiene los 2 alelos iguales; heterocigoto, si tiene los 2 alelos distintos o hemicigoto, si tiene un único alelo para los genes de los cromosomas X e Y en un varón. Un haplotipo es una combinación de alelos, o conjunto de variaciones del DNA, que se heredan conjuntamente debido a su localización cromosómica muy próxima.
3. ENFERMEDADES GENÉTICAS Y PATRONES DE HERENCIA
Una enfermedad genética está causada por una alteración del genoma y puede ser hereditaria o no. Si el gen alterado está presente en los gametos (óvulos y espermatozoides) de la línea germinal la enfermedad será hereditaria, en cambio, si altera únicamente las células somáticas no será hereditaria, como ocurre en la mayoría de los cánceres que son causados por mutaciones adquiridas (somáticas).
Las enfermedades genéticas hereditarias se pueden clasificar en 4 tipos según el tipo de alteración genética que las causa y su patrón de herencia: cromosómicas, monogénicas, mitocondriales y poligénicas.
Las anomalías cromosómicas se presentan en aproximadamente el 0.4% de la población general y pueden afectar el número o la estructura de los cromosomas. La mayoría de las anomalías cromosómicas se generan durante la meiosis. A pesar de que es posible heredar algunos tipos de trastornos cromosómicos, la mayoría no se transmiten de una generación a la siguiente sino que son de novo (no heredadas).
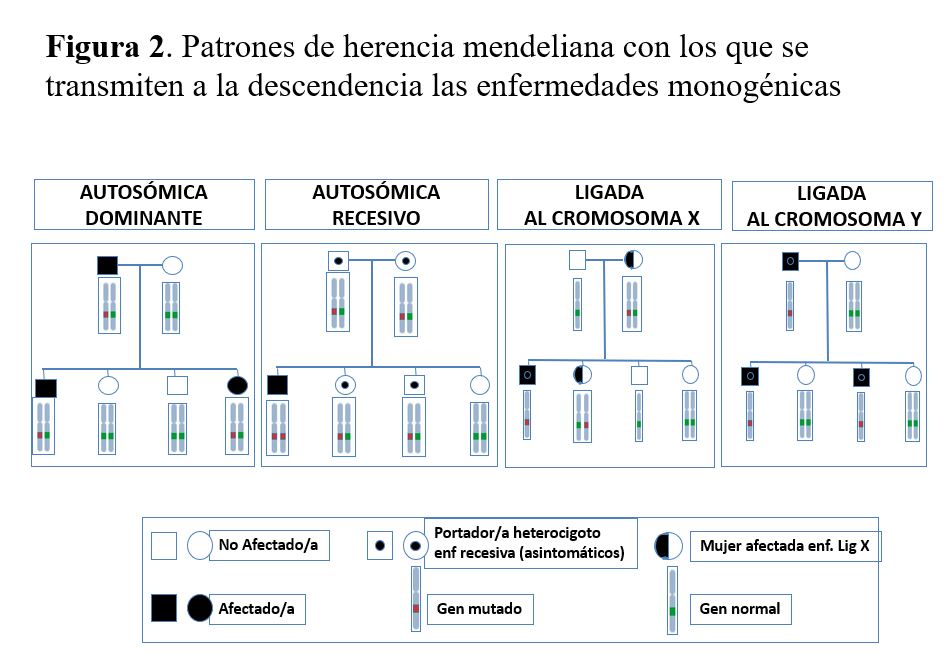
Las enfermedades monogénicas, también denominadas mendelianas, son mayoritariamente enfermedades raras y están causadas por una variante patogénica en un único gen de los aproximadamente 20.000 genes que contiene nuestro genoma. Se transmiten a la descendencia siguiendo los patrones de herencia mendeliana: autosómico dominante, autosómico recesivo, ligado al cromosoma X y ligado al cromosoma Y (Figura 2). Los genes implicados en las enfermedades autosómicas están localizados en uno de los 22 pares de autosomas, mientras que los ligados al sexo se encuentran en el cromosoma X o Y.
En las enfermedades con herencia autosómica dominante, como la PQRAD, la presencia de una sola copia del gen mutado es suficiente para que la enfermedad se manifieste, de manera que los individuos afectados presentan la mutación en heterocigosis. Las principales características de este patrón de herencia son: la transmisión vertical, en la que cada paciente tiene el padre o la madre también afectado por la enfermedad (salvo en mutaciones de novo); existe el mismo riesgo para ambos sexos a padecer o transmitir la enfermedad y una probabilidad del 50% de que los descendientes hereden la copia del gen mutado y estén afectados por la enfermedad.
En las enfermedades con herencia autosómica recesiva, como la nefronoptisis, únicamente los individuos con las dos copias del gen mutadas manifiestan la enfermedad. Las principales características son: los progenitores del individuo afectado son portadores heterocigotos de la mutación y generalmente asintomáticos; transmisión horizontal, es decir, si aparece más de un afectado en la familia suelen ser hermanos; el riesgo de recurrencia de la enfermedad en cada hermano del probando es del 25% y los varones y mujeres tienen la misma probabilidad de estar afectados. Las enfermedades recesivas suelen manifestarse más precozmente que las dominantes, incluso en la etapa prenatal o en la infancia y adolescencia y presentan penetrancia completa. Otro factor a considerar en estas enfermedades es la consanguinidad y la endogamia, las cuales aumentan la probabilidad de que los miembros de una pareja sean portadores de una mutación en el mismo gen.
Las enfermedades de herencia ligada al cromosoma X, como la enfermedad de Fabry, generalmente se manifiestan de forma más grave en los hombres que en las mujeres. Esto es debido a que los varones son hemicigotos para los genes del cromosoma X (sólo tienen una copia) y a que en las mujeres con 2 cromosomas X se produce el fenómeno de la inactivación del cromosoma X durante la primera semana del desarrollo. El X inactivo puede ser el paterno o el materno y se inactiva de forma aleatoria pero permanente. Si uno de los cromosomas X está mutado, la proporción de células con el gen mutado activo es variable. Este fenómeno explica la variabilidad de la severidad que pueden presentar las mujeres para estas enfermedades e indica que la designación de mujer afectada de una enfermedad ligada al cromosoma X puede generar confusión, ya que las mujeres también están afectadas aunque con grado muy variable. Otras características de las enfermedades con herencia ligada al X son: el varón transmite la variante patogénica a todas sus hijas que la presentarán en heterocigosis; no hay transmisión varón-varón de la enfermedad; los varones afectados estarán emparentados siempre a través de mujeres.
Por último, las enfermedades de herencia ligada al cromosoma Y que sólo afectan a los hombres, de manera que se transmiten del padre afectado a todos sus hijos varones y a ninguna de sus hijas. No existe ninguna ERH con patrón de herencia ligado al cromosoma Y.
Recientemente, se ha acuñado el término de herencia semidominante para designar algunos genes que pueden estar asociados tanto a un patrón de herencia autosómico dominante como recesivo. En este caso los individuos con una variante patogénica en heterocigosis presentan un fenotipo más leve en comparación con los que presentan variantes patogénicas bialélicas (en las 2 copias del gen en homocigosis o heterocigosis compuesta) que presentan una forma más grave y de aparición más temprana de la enfermedad. Un ejemplo serían los genes COL4A3 y COL4A4, que cuando presentan una variante patogénica en heterocigosis causan el síndrome de Alport autosómico dominante de debut en el adulto con presentación y penetrancia variables de la microhematuria, proteinuria, enfermedad renal crónica y sordera, mientras que cuando estos genes tienen sus dos copias mutadas causan el síndrome de Alport autosómico recesivo con una presentación clínica más grave y de debut generalmente en la edad pediátrica.
Las enfermedades mitocondriales con herencia materna están causadas por una variante patogénica en el DNA mitocondrial (mtDNA), como el síndrome de MELAS que puede cursar con afectación renal. El mtDNA es un pequeño cromosoma circular que contiene 37 genes que se encuentra dentro de las mitocondrias. Las mitocondrias son orgánulos encargados de la producción de energía. Las mitocondrias y, por tanto, el mtDNA se transmiten por vía materna, ya que es el oocito el que aporta miles de copias de mtDNA al cigoto. Cada célula tiene entre 1.000-100.000 moléculas de mtDNA. El mtDNA tiene una tasa de mutación 10-20 veces superior al DNA nuclear y es común que coexistan en una célula moléculas de mtDNA mutadas y no mutadas, lo que se denomina heteroplasmia. El fenotipo de un individuo heteroplásmico depende del porcentaje de mtDNA mutado. Por ello, las enfermedades debidas a mutación en el mtDNA tienen una gran variabilidad clínica. Una mutación concreta en el mtDNA puedo producir fenotipos muy variables y mutaciones diferentes pueden producir síntomas similares.
Por último, las enfermedades más comunes son las enfermedades poligénicas también denominadas multifactoriales o complejas, como la diabetes y la hipertensión, que no son monogénicas como su nombre indica. Están causadas tanto por factores de predisposición genéticos como ambientales, es decir, que varios alelos de genes diferentes proporcionan un riesgo genético o predisposición individual a su desarrollo, que sólo se manifiestan en ciertas condiciones ambientales. Estas enfermedades tienen un patrón de herencia complejo y muestran una agregación familiar mucho menor que las monogénicas. Para cada variante genética la correlación genotipo-fenotipo es muy baja y usualmente sólo puede asignarse un riesgo relativo a una determinada variante genética. Las enfermedades poligénicas generalmente debutan en la edad adulta y son mucho más frecuentes en la población que las enfermedades monogénicas.
4. MÉTODOS DE DIAGNÓSTICO GENÉTICO
El diagnóstico genético consiste en analizar el material genético, generalmente DNA genómico, obtenido de un paciente con sospecha clínica de estar afectado de una enfermedad genética con el fin de identificar la alteración genética que causa la enfermedad.
Las metodologías que se utilizan para el diagnóstico genético son muy variadas, permitiendo desde el estudio de cromosomas hasta el análisis del cambio de un único nucleótido. Las alteraciones a nivel cromosómico se analizan con técnicas de citogenética y las alteraciones génicas se analizan mediante las técnicas de genética molecular [14].
4.1. Análisis cromosómico
El análisis cromosómico en pacientes con enfermedad renal está principalmente indicado en pacientes con cuadros sindrómicos.
Las alteraciones cromosómicas se han analizado durante años con el cariotipo tradicional que continua utilizándose. Permite detectar mutaciones cromosómicas numéricas (poliploidía, haploidía, aneuploidía) y algunas mutaciones cromosómicas estructurales (grandes deleciones y duplicaciones, inversiones, translocaciones). Sin embargo, esta técnica tiene una baja resolución (5-10 millones de pares de bases (Mb)), es laboriosa y necesita 2-3 semanas para el cultivo celular (linfocitos, amniocitos,etc.).
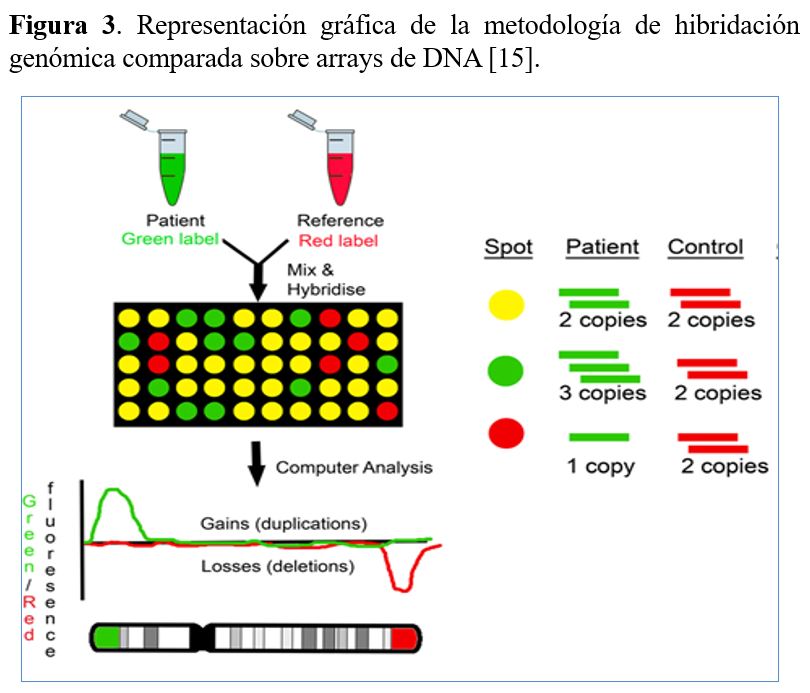
En los últimos años el cariotipo tradicional ha sido ampliamente remplazado por el cariotipo molecular realizado con la metodología de hibridación genómica comparada sobre arrays de DNA conocida como aCGH (Array Comparative Genomic Hybridization). El análisis se lleva a cabo mediante la co-hibridación del DNA del paciente y un DNA control (normal) marcados con fluorocromos distintos en un soporte sólido (array) que contiene fragmentos de DNA inmovilizados (sondas) diseñados específicamente para la detección de anomalías a lo largo de todo el genoma (Figura 3). Cantidades equimolares del DNA del paciente (marcado con un fluorocromo verde) y el control (marcado con un fluorocromo rojo) compiten para unirse (hibridar) con las sondas del array de manera que si no hay ninguna alteración hibridan por igual (amarillo) y si hay una deleción (pérdida de DNA) o una duplicación (ganancia de DNA) en el paciente se une preferentemente el DNA del control o del paciente, respectivamente. Esta metolología permite detectar deleciones y duplicaciones, qué desde la introducción de los aCGH, se las denomina también variantes de número de copia o CNVs (Copy Number Variants). La resolución del array (capacidad de detectar anomalías más pequeñas) depende del número de sondas del array. Al aumentar el número de sondas, aumenta la cobertura del genoma y disminuye el espacio entre las sondas, lo que posibilita identificar CNVs más pequeñas, incluso del nivel de un exón.
Las principales ventajas del aCGH con respecto al cariotipo tradicional son:
1) Alta resolución, que permite detectar microdeleciones y microduplicaciones.
2) Obtención de resultados en prácticamente todos los casos, evitando los fallos de cultivo celular.
3) Mayor rapidez, ya que al no requerir cultivo celular de la muestra permite obtener resultados en 7 días.
4) Análisis bioinformático de los datos de hibridación, que evita variación en la interpretación de los resultados entre profesionales.
El inconveniente del aCGH es que no permite detectar alteraciones cromosómicas estructurales sin cambios en el número de copia, como las translocaciones cromosómicas equilibradas, inversiones, disomías uniparentales y está limitado para la detección de mosaicismo.
La interpretación clínica precisa de los resultados del aCGH es el principal desafío, ya que se desconoce el significado clínico de muchas de las CNVs que se detectan. Por ello, es imprescindible que el clínico que solicita el estudio genético proporcione información clínica detallada al laboratorio para poder realizar una correcta interpretación clínica de los resultados genéticos.
4.2. Análisis genético-molecular
La aproximación más utilizada para el diagnóstico genético de las ERH es el análisis mutacional del gen o genes candidatos a causar la ERH. El objetivo es identificar la variante patogénica (mutación) que causa la enfermedad en el paciente.
En los casos familiares generalmente se realiza el análisis genético completo del gen o conjunto de genes al familiar más gravemente afectado (caso índice o probando) y en el resto de familiares únicamente se realiza un estudio genético secundario dirigido a determinar presencia/ausencia de la variante patogénica identificada en el caso índice, lo que permite confirmar o descartar la enfermedad en sus familiares.
La principal limitación del análisis directo es qué en caso de no identificar ninguna variante patogénica, el resultado negativo del estudio genético no confirma ni descarta que el paciente esté afectado de una enfermedad monogénica, ya que todas las metodologías tienen una sensibilidad inferior al 100%.
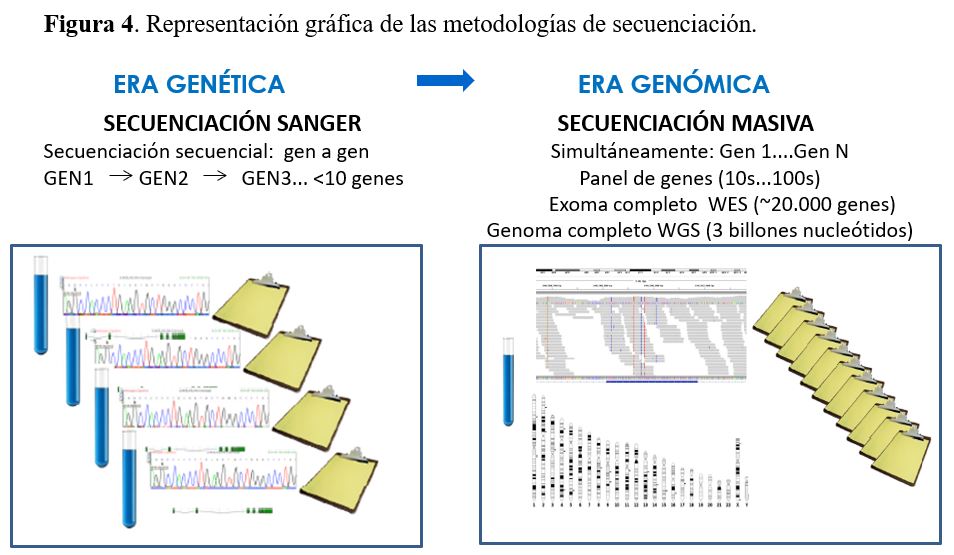
La metodología más ampliamente utilizada en el análisis mutacional es la secuenciación del DNA que puede realizarse con distintos métodos (Figura 4) y (Figura 5).
4.2.1. Secuenciación Sanger
Es el método clásico de secuenciación del DNA con el que se llevó a cabo el Proyecto Genoma Humano que duró más de 10 años (1990-2003) y logró secuenciar el genoma completo de un individuo y costó 3 billones de dólares.
La secuenciación Sanger es un método muy sensible para la identificación de mutaciones, pero en cada experimento sólo permite secuenciar un solo fragmento de DNA de una longitud máxima de unos 1000 nucleótidos. Por ello, para el análisis mutacional de un gen es necesario secuenciar independientemente cada uno de los exones del gen (partes que codifican para proteína del gen). Esta metodología resulta muy laboriosa para enfermedades causadas por genes con gran número de exones (p. ej. gen PKHD1 con 67 exones) y/o enfermedades con heterogeneidad genética (con múltiples genes causales), ya que si no se identifica la mutación en el primero gen candidato, se debe secuenciar el segundo gen candidato, el tercero... hasta llegar a identificar la mutación. La selección del primer gen candidato a secuenciar viene determinada por las características clínicas del paciente y por el porcentaje de casos que explica cada uno de los genes (p. ej. en la PQRAD el análisis secuencial generalmente se empieza secuenciando el gen PKD1 causante del 78% de los casos, el segundo gen PKD2 causante del 15%, seguido de GANAB causante del 0,3% de los casos, DNAJB11 causante del 0,1% de los casos y en aproximadamente el 7% de los casos no se encuentra mutación en ninguno de estos 4 genes).
Actualmente, la secuenciación de Sanger continúa siendo el método utilizado en determinados casos:
1) Diagnóstico de una enfermedad monogénica que no presenta heterogeneidad genética y cuyo gen causante está constituido por pocos exones. Un ejemplo sería la enfermedad de Fabry causada por el gen GLA con sólo 7 exones.
2) Estudio de familiares de un caso índice, cuya mutación ya ha sido identificada, para confirmar/descartar la mutación identificada en el caso índice.
3) Confirmar los resultados obtenidos por técnicas de secuenciación masiva.
La secuenciación de Sanger únicamente permite identificar variantes de secuencia puntuales, aquellas que alteran un nucleótido o unos pocos. Por ello, esta técnica debe complementarse con la técnica denominada multiplex ligation-dependent probe amplification (MLPA) que permite detectar deleciones y duplicaciones de un exón, múltiples exones o un gen completo.
4.2.2. Secuenciación masiva
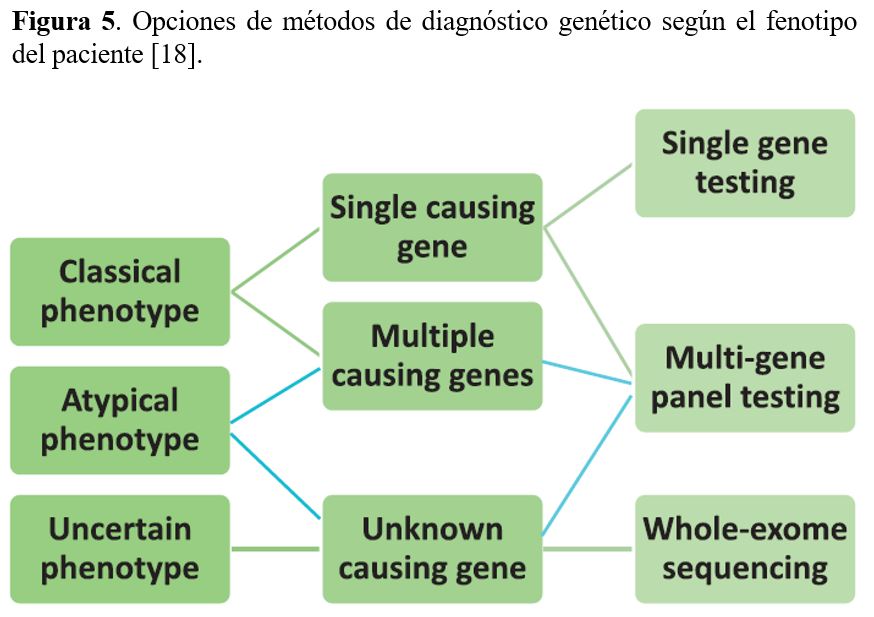
La secuenciación masiva o Next Generation Sequencing (NGS) es la técnica de secuenciación más utilizada actualmente para el diagnóstico genético de las ERH. Permite la secuenciación simultánea de todos los genes relevantes para un determinado fenotipo a un precio y tiempo de respuesta mucho más reducido que con el método clásico de secuenciación de Sanger y con una sensibilidad del mismo orden. Además, la NGS permite la detección de todo tipo de variantes genéticas, desde variantes de un solo nucleótido hasta CNVs, aunque la sensibilidad para la detección de CNVs es menor. La NGS puede realizarse a distintos niveles: a) paneles de genes, b) exoma completo y c) genoma completo.
a) La secuenciación masiva de un panel de genes se centra en el análisis exhaustivo de un número determinado de genes asociados con una enfermedad específica o grupo de enfermedades. Los paneles de genes NGS utilizan el enriquecimiento dirigido de genes seleccionados para proporcionar una secuenciación rápida y económica con una cobertura más alta que la lograda con exoma completo o genoma completo. Es la metodología de elección en muchos laboratorios de diagnóstico. La eficiencia diagnóstica de un panel de genes depende totalmente del diseño del panel, de la técnica de enriquecimiento de las regiones de interés y de la profundidad de lectura (depth of coverage) a la que se secuencie. Algunos paneles contienen los genes específicos para un determinado fenotipo [16] y otros paneles pueden ser muy amplios y contener los genes causantes de todas las ERH [17].
A nivel técnico, las principales ventajas de la secuenciación de un panel de genes con respecto la secuenciación del exoma completo son las siguientes:
1) Permite una mayor eficiencia en la identificación de CNVs, mutaciones complejas (indels), mutaciones en regiones genómicas complejas (pej PKD1 con 6 pseudogenes, regiones ricas en GCs, ...) e identificar mosaicismo. Esta mayor eficiencia es debida a que se obtiene una profundidad de lectura (depth of coverage, el número de veces que se lee cada nucleótido del gen) muy elevada (>200x).
2) Se detectan un número mucho menor de variantes genéticas candidatas a ser la causa de la patología que presenta el paciente, lo que implica que el análisis de los resultados es más rápido y menos complejo.
3) Evita identificar variantes en genes implicados en otras patologías (incidental/secondary findings).
Las principales desventajas de la secuenciación de un panel de genes con respecto al exoma:
1) No permite identificar mutaciones en genes no incluidos en el panel.
2) Requiere una actualización periódica del diseño del panel para incluir los nuevos genes descubiertos asociados a la patología de estudio.
3) Capacidad más baja de reanálisis de los datos de secuenciación.
b) La secuenciación del exoma completo o Whole Exome Sequencing (WES) es un enfoque más global que el panel de genes, ya que analiza la mayoría de exones de la mayoría de genes que conforman el genoma humano (~20.000 genes) [4]. En muchos laboratorios se usa el WES cuando la NGS de un panel de genes no ha identificado ninguna causa monogénica de la enfermedad. Las principales ventajas del WES con respecto a un panel:
1) Permite detectar mutaciones en la gran mayoría de genes del genoma.
2) Posibilita descubrir un nuevo gen candidato a causar una enfermedad, no previamente descrito en la literatura, aunque el nuevo gen no tendrá valor diagnóstico hasta que se identifique como causante en múltiples pacientes con clínica similar y/o existan estudios funcionales que lo avalen.
3) Permite un reanálisis periódico de los datos de secuenciación incorporando nuevos métodos bioinformáticos y el conocimiento científico de nuevos genes causantes de enfermedad.
Las principales desventajas de la secuenciación del WES:
1) Sensibilidad limitada de regiones genómicas complejas que pueden ser clínicamente relevantes (p. ej. la región duplicada del gen PKD1 con 6 pseudogenes, regiones ricas en GCs,...).
2) Probabilidad más elevada de identificar resultados no solicitados (incidental findings).
3) Gran cantidad de variantes para interpretar, lo que dificulta la identificación de la variante patogénica causante de la enfermedad, y de datos para almacenar.
c) La secuenciación del genoma completo o Whole Genome Sequencing (WGS) analiza tanto los exones como los intrones (regiones que no codifican para proteína) de todos los genes del genoma, así como las regiones intergénicas. No requiere ninguna técnica de enriquecimiento ya que se secuencia todo el genoma. Hoy en día, WGS se usa mayoritariamente para proyectos de investigación o para casos muy complejos en los que el WES ha resultado negativo y hay una muy clara sospecha de que la causa de la enfermedad es genética. Tiene la ventaja que puede detectar determinados reordenamientos cromosómicos que no se pueden detectar ni con paneles ni con WES.
Los costes de secuenciación se van reduciendo progresivamente, pero la cantidad de datos de secuencia generados se está expandiendo y, en consecuencia, el análisis y el almacenamiento de datos se están convirtiendo en una de las proporciones más grandes del coste real de la secuenciación. La bioinformática que es la disciplina encargada del análisis y la gestión de esta gran cantidad de datos se ha convertido en una parte esencial del diagnóstico genético.
El objetivo del diagnóstico genético es identificar la variante causante de la enfermedad monogénica de entre los miles de variantes de secuencia identificadas en cualquier paciente con respecto a la secuencia del genoma de referencia. Por ello, será imprescindible un filtrado de las variantes que tiene el paciente, para descartar las que son benignas/probablemente benignas (variantes frecuentes en la población general, localizadas en regiones intrónicas o intergénicas que no codifican para proteína, ...) y analizar con detalle el subgrupo de variantes candidatas a ser patogénicas. Este filtrado se realiza teniendo en cuenta la clasificación de las variantes en función de su patogenicidad (ver apartado 5.2.1).
La principal dificultad de la NGS es la interpretación clínica de los datos de secuenciación. Por ello, es esencial la precisa descripción de las características clínicas del paciente en estudio y de sus familiares, que nos indicarán cuales de las variantes potencialmente patogénicas detectadas pueden tener un significado clínico relevante. También es de gran ayuda disponer de la muestra e información clínica de miembros de la familia que permitan el análisis de segregación de las principales variantes patogénicas candidatas identificadas en el caso índice (comprobando que los familiares enfermos tengan la variante patogénica y los sanos no la tengan). Finalmente, la experiencia de genetistas y clínicos formados específicamente en ERH resultan esenciales para un diagnóstico genético fiable y de calidad.
4.2.3. Otras técnicas de diagnóstico genético-molecular:
Algunas variantes patogénicas no se pueden identificar mediante ninguna de las técnicas hasta ahora mencionadas, como por ejemplo la principal variante patogénica identificada en el gen MUC1 causante de nefropatía tubulointersticial autosómica dominante (ADTKD-MUC1). Se trata de la variante MUC1 (NM_001204286.1): c.428dupC p.(Ala144Serfs*86) que consiste en la duplicación de una única citosina en un homopolímero de 7 citosinas en una repetición en tándem de número variable de copias (VNTR) (GC-rich 60-base VNTR). Esta variante al estar localizada en un VNTR se analiza con una técnica específicamente diseñada basada en minisecuenciación o SNaPshot [19], o por espectrometría de masas [20].
El análisis de ligamiento genético es una aproximación indirecta para realizar el diagnóstico genético de determinadas ERH, sólo en los casos familiares y siempre y cuando el diagnóstico clínico de la ERH en la familia sea 100% certero [21]. Consiste en determinar y comparar los haplotipos (combinación de alelos de varios marcadores genéticos situados en la región del gen) de los distintos familiares con el fin de determinar el haplotipo de riesgo que segrega con la enfermedad en una determinada familia con una enfermedad monogénica. El haplotipo de riesgo es el que comparten todos los familiares afectados y que no presenta ninguno de los familiares sanos. Únicamente se puede realizar esta aproximación si se dispone de muestra de DNA e información clínica de múltiples miembros de una misma familia de distintas generaciones, tanto afectados como no afectados por la enfermedad en estudio. Esta aproximación indirecta no es aplicable en enfermedades con elevada heterogeneidad genética. Puede ser útil en determinados casos en los que no se ha identificado la variante patogénica mediante el análisis directo y también se utiliza en el test genético preimplantacional.
5. NOMENCLATURA Y CLASIFICACIÓN DE LAS VARIANTES DE SECUENCIA
5.1. Nomenclatura
Las variantes de secuencia se nombran utilizando una nomenclatura uniforme y estandarizada que permite una designación inequívoca de cada variante y hace factible un uso compartido de la información genómica. Las guías de nomenclatura son establecidas por la Human Genome Variation Society (HGVS) y se van actualizando periódicamente. Estas guías están disponibles en internet (http://www.hgvs.org/mutnomen/).
En los informes de diagnóstico genético se reportan únicamente las variantes causantes o asociadas a la clínica que presenta el paciente, utilizando la nomenclatura HGVS válida en el momento del informe. Se debe incluir la secuencia de referencia del gen (p. ej. para PKD1 NM_001009944.3) y la nomenclatura de la variante tanto a nivel de secuencia de DNA codificante (p. ej. c.8311G>A) como de proteína (p. ej. p.(Glu2771Lys)) generalmente entre paréntesis para indicar que es un efecto predicho. También debe indicarse si la variante se ha identificado en el paciente en heterocigosis, en homocigosis o en hemicigosis.
5.2. Clasificación
Las variantes de secuencia identificadas en un individuo con respecto al genoma de referencia pueden clasificarse según distintos criterios:
a) Según su patogenicidad:
La clasificación de la patogenicidad de las variantes de secuencia es una de las tareas más relevantes del diagnóstico genético. Identificar una variante patogénica o probablemente patogénica en un gen mendeliano permite definir un diagnóstico etiológico preciso que puede ser determinante en el manejo del paciente.
En el año 2015, el American College of Medical Genetics (ACMG) y la Association for Molecular Pathology (AMP) describió unas guías para la interpretación y clasificación sistemática de las variantes de secuencia en base a su probabilidad de causar una determinada enfermedad monogénica [22]. Actualmente, estas recomendaciones son ampliamente usadas en los laboratorios de diagnóstico genético de todo el mundo.
Estas guías permiten clasificar las variantes de secuencia en 5 categorías:
1. Patogénica: causante de enfermedad, con valor diagnóstico para la enfermedad asociada.
2. Probablemente patogénica: probablemente causante de enfermedad, a la práctica con valor diagnóstico para la enfermedad asociada.
3. Variante de significado clínico incierto (VUS): con causalidad no definida con la información disponible actualmente. Muchas variantes se clasifican en esta categoría lo que genera una confusión significativa en la práctica clínica.
4. Probablemente benigna: probablemente no causante de enfermedad.
5. Benigna: no causante de enfermedad.
Se consideran causantes de una enfermedad las variantes que resultan clasificadas en las 2 primeras categorías: "patogénica" y "probablemente patogénica". El resto de categorías no tienen valor diagnóstico. No obstante, las variantes de significado incierto o VUS (Variants of Unknown Significance) pueden ser reportadas en un informe de diagnóstico genético principalmente cuando no se ha identificado ninguna variante patogénica/probablemente patogénica y si se localizan en un gen que se ha asociado al fenotipo que presenta el paciente. Muchas de estas variantes VUS se van reclasificando en probablemente patogénicas o probablemente benignas a medida que se dispone de más evidencias.
Para clasificar las variantes las guías de la ACMG/AMP evalúan diferentes criterios de evidencia:
1) Características inherentes a cada variante p. ej. variantes truncantes (ver apartado c) en muchos genes mendelianos implican una fuerte evidencia de patogenicidad.
2) Frecuencia de cada variante en bases de datos poblacionales de referencia como p. ej. Genome Aggregation Database (gnomAD). Las variantes patogénicas generalmente no figuran en estas bases de datos poblacionales o tienen frecuencias poblacionales extremadamente bajas.
3) Predicciones de patogenicidad de las variantes de cambio de aminoácido (missense) obtenidas con múltiples algoritmos bioinformáticos disponibles online (p. ej. Mutation Taster, Polyphen, SIFT, Mutation Assessor) que evalúan la probabilidad de que una variante sea o no patogénica.
4) Correlación genotipo-fenotipo, es decir, que el fenotipo o los antecedentes familiares del paciente correlacionen con las manifestaciones clínicas atribuidas al gen que presenta la variante candidata.
5) Patrón de herencia del gen, p. ej. si el gen es recesivo debe existir otra variante candidata en el mismo gen en trans (es decir, en la otra cadena de cromatina).
6) Segregación familiar de la variante: si la variante se localiza en un gen autosómico dominante, comprobar que los familiares afectos presentan la variante y los sanos no la presentan. Si el paciente es un caso de novo de la enfermedad, comprobar que los padres no presentan la variante.
7) Artículos científicos en los que se describe la variante con datos funcionales que indican una alteración de la proteína resultante y/o información clínica de los pacientes en que se ha identificado la variante candidata.
8) Bases de datos de correlación genotipo-fenotipo como ClinVar o Human Mutation Database (HMDB) o de genes específicos como la PKD Mayo database para los genes PKD1 y PKD2; la LOVD para los genes COL4A3 y COL4A4, en las que generalmente figura la interpretación clínica de la variante, si ésta ha sido descrita en la literatura científica.
Estos criterios se combinan de acuerdo con unas reglas de puntuación [22] para elegir una clasificación de cada variante en una de las 5 categorías arriba mencionadas.
b) Según el tipo de alteración de nucleótido:
Como ya se ha comentado, las variantes de secuencia pueden ser puntuales, es decir, que afectan a un solo nucleótido (SNVs, single nucleotide variants) o unos pocos nucleótidos, o bien alterar una porción significativa del DNA, en este último caso se denominan variantes estructurales o CNVs. Una de las principales bases de datos de mutaciones The Human Gene Mutation Database las clasifica según el tipo de alteración de nucleótidos (Tabla 1).
c) Según el potencial efecto que la variante causa en la proteína:
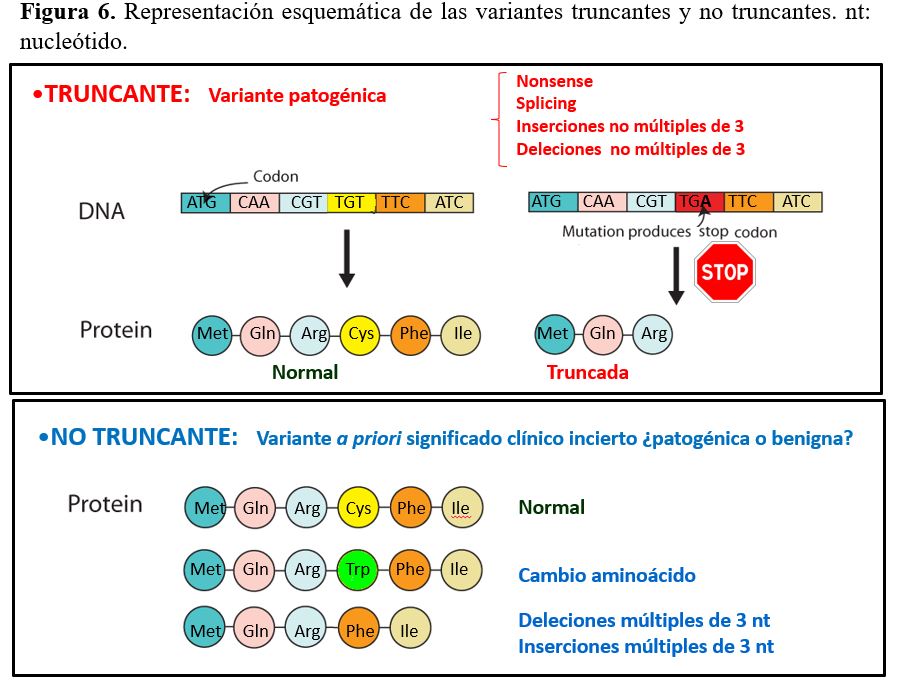
Las variantes de secuencia también se pueden clasificar en truncantes o no truncantes (Figura 6). Las variantes truncantes dan lugar a la creación de codón de terminación de la traducción proteica prematuro resultando en una proteína truncada (de tamaño inferior a la proteína normal).
Las variantes truncantes pueden ser de distintos tipos:
Nonsense (sin sentido): cambio de nucleótido que da lugar a la creación de un codón de stop (TAG, TAA o TGA), que termina prematuramente la traducción produciendo una proteína truncada.
Frameshift (cambio de pauta de lectura): Inserción o deleción de uno o varios nucleótidos cuyo número no es múltiplo de 3, lo que genera un codón stop prematuro.
Splicing: que conlleva el salto de un exón (exon skipping) o fragmento de exón o inserción de una parte de un intrón en la secuencia codificante con un número de nucleótidos no múltiple de 3.
Las variantes no truncantes no alteran la pauta de traducción proteica (in frame). En este grupo se incluyen las variantes de tipo:
Missense (cambio de aminoácido): cambio de nucleótido que da lugar a un cambio de aminoácido.
Deleciones o inserciones en pauta (in frame): de un número de nucleótidos múltiplo de tres.
Splicing: que conlleva el salto de un exón (exon skipping) o fragmento de exón o inserción de una parte de un intrón en la secuencia codificante con un número de nucleótidos múltiple de 3.
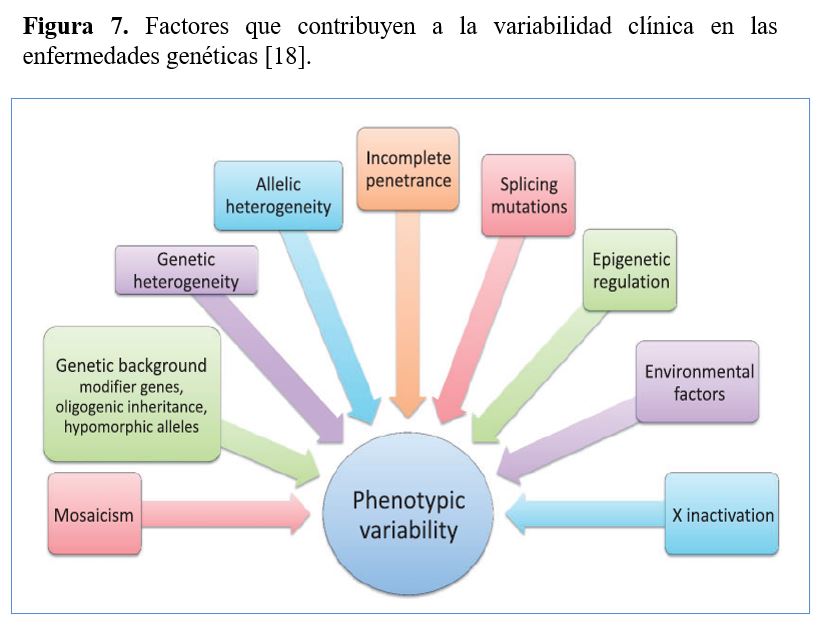
6. FACTORES QUE CONTRIBUYEN A LA VARIABILIDAD FENOTÍPICA DE LAS ERH
A día de hoy, todavía se desconoce porque pacientes con la misma ERH pueden tener presentaciones clínicas muy distintas. Algunos de los factores que pueden contribuir a esta elevada variabilidad fenotípica de las ERH son los siguientes (Figura 7)[18]:
a) Heterogeneidad genética y alélica
La heterogeneidad genética que presentan varias ERH (que aunque siendo monogénicas el gen mutado que las puede causar no es único) puede explicar parte de su variabilidad fenotípica ya que según el gen causante el fenotipo puede diferir. Un claro ejemplo es la PQRAD, donde los pacientes con mutaciones de PKD1 alcanzan TRS unos 20 años antes que aquellos con mutaciones PKD2.
La heterogeneidad alélica se refiere a las distintas mutaciones de un mismo gen (alelos), que pueden dar lugar al mismo fenotipo o diferentes fenotipos. Los tipos de mutación se correlacionan con la gravedad de la enfermedad en varias ERH. Un ejemplo son los pacientes portadores de dos mutaciones truncantes en el gen PKHD1 que presentan una forma muy grave de poliquistosis renal autosómica recesiva (PQRAR), letal en muchos casos, mientras que la presencia de al menos una mutación missense está asociada a una forma menos grave con mejor pronóstico. En general, las correlaciones genotipo-fenotipo son más débiles para los trastornos monogénicos autosómicos dominantes que para las enfermedades autosómicas recesivas.
b) Penetrancia y Expresividad
La penetrancia se define como la probabilidad de manifestar un fenotipo cuando se tiene un genotipo determinado. Si el genotipo siempre se expresa la penetrancia es completa, es decir, que tener la mutación es sinónimo de manifestar la enfermedad. En cambio, si se puede presentar la mutación y no estar afectado por la enfermedad la penetrancia es incompleta (inferior al 100%). Debe tenerse en cuenta que la penetrancia depende de la edad. En general, para la mayoría de enfermedades recesivas la penetrancia es completa mientras que para las enfermedades dominantes la penetrancia es incompleta (al menos hasta cierta edad). Un ejemplo de penetrancia incompleta son los pacientes con una mutación en heterocigosis en los genes COL4A3 o COL4A4 que pueden estar completamente asintomáticos o presentar sólo hematuria intermitente.
La expresividad es el grado de manifestación de un fenotipo. Se considera que la expresividad es variable cuando la presentación de un fenotipo varía entre individuos poseedores del mismo genotipo. Esta variabilidad fenotípica puede ser tanto interfamiliar, entre individuos no relacionados, como intrafamiliar. Cabe destacar que muchas enfermedades presentan una expresividad variable con la edad, de manera que un fenotipo muy leve en la infancia no excluye un desarrollo moderado o grave de la enfermedad.
La penetrancia incompleta y la expresividad variable pueden explicarse sobre la base de la existencia de genes modificadores de la gravedad de la enfermedad y por la influencia de factores ambientales, que modulan el efecto del gen principal causante de la enfermedad.
c) Herencia oligogénica y genes modificadores
En la herencia oligogénica el fenotipo de la enfermedad está determinado por variantes patogénicas en unos pocos genes. Esta herencia se ha sugerido, aunque no claramente demostrado, en algunas ERH como el síndrome de Bardet-Biedl [23].
El concepto de factor genético modificador se utiliza para una variante de secuencia que por sí sola no causa la enfermedad pero contribuye al fenotipo de la enfermedad. Las variantes genéticas modificadoras pueden localizarse en el gen causante de la ERH o en otros genes denominados genes modificadores, ya que no causan la enfermedad pero modifican su fenotipo. Es probable que los genes modificadores tengan un papel relevante para explicar la variabilidad clínica intrafamiliar, especialmente en las enfermedades autosómicas dominantes. En algunos casos de PQRAD, NTAD y el síndrome de Alport autosómico dominante existe una enorme variabilidad clínica intrafamiliar con más de 30 años de diferencia en la edad de inicio de TRS en diferentes miembros de la familia.
Estudios de familias con PQRAD con algún familiar con un debut muy temprano de la enfermedad han identificado que además de la mutación PKD1 familiar presenta en el otro alelo una variante PKD1 hipomórfica o de pentrancia incompleta. Las variantes hipomórficas por sí solas no causan enfermedad, aunque pueden causar alguna manifestación clínica leve (p.ej. unos pocos quistes), pero en trans (en el otro alelo) con otra variante hipomórfica o patogénica dan lugar a un fenotipo variable que puede ser muy grave y de debut muy temprano [24] [25].
d) Mosaicismo
El mosaicismo se define como la coexistencia de dos o más poblaciones de células con distintos genotipos (con mutación y sin mutación) en un individuo originado de un único cigoto. Existen 3 tipos de mosaicismo: somático, germinal y gonosómico, según si afecta a las células somáticas, a las germinales o a los dos tipos. Dependiendo de la expresión del alelo mutado, tanto en términos de porcentaje como de expresión específica de órganos, surgen diferentes fenotipos. El mosaicismo puede explicar la expresión clínica leve de la enfermedad en pacientes que presentan una ERH autosómica dominante de novo (sin antecedentes familiares). También debe tenerse en cuenta en el asesoramiento genético reproductivo de los padres asintomáticos de un caso de novo de una ERH autosómica dominante. Se debería informar a los padres que tienen un riesgo muy bajo, pero no nulo, de tener otro hijo afectado, ya que uno de los progenitores asintomáticos podría presentar mosaicismo germinal (coexistencia de óvulos o espermatozoides mutados y no mutados).
e) Inactivación del cromosoma X
El nivel de expresión de los genes que produce una sola copia del cromosoma X es la dosis normal para un ser humano. Por ello, en las mujeres se produce la inactivación de un cromosoma X mediante un proceso aleatorio que pasa por separado en células individuales durante el desarrollo embrionario. Todas las células descendientes de cada una de estas células embrionarias, mantendrá el mismo patrón de inactivación del cromosoma X. El cromosoma X inactivado está silenciado y sus genes no se traducen a proteína. La elección de cuál de los dos cromosomas X se inactiva es aleatoria.
En el caso que exista una mutación en un gen del cromosoma X en una mujer, si la inactivación del cromosoma X en los órganos diana de la enfermedad ha sido aleatoria, la mujer presentará unas manifestaciones clínicas menos graves que los varones con la misma mutación. No obstante, en ciertos casos la inactivación no es al azar sino que se inclina hacia el cromosoma X sin mutación o con mutación. Si hay un alto porcentaje de células con el cromosoma X sin mutación inactivado, la enfermedad es más grave de lo que cabría esperar en una mujer. Si por el contrario hay un alto porcentaje de células con el cromosoma X mutado inactivado la mujer será prácticamente asintomática. Este fenómeno es específico de cada célula, por lo que los niveles de inactivación del X en un tipo de celular u órgano no se pueden extrapolar a otros órganos.
f) Mutaciones de splicing
El splicing es un mecanismo mediante el cual se eliminan los intrones (regiones del gen que no codifican proteina) y se unen los exones (regiones del gen que codifican proteína) para dar lugar al RNA mensajero (mRNA) o transcrito maduro que sirve como plantilla para la síntesis de una proteína específica.
Existen unas secuencias canónicas de splicing que son esenciales para que el este mecanismo se produzca correctamente. Estas secuencias son la secuencia donadora o 5´ splice site (GT) y la secuencia aceptadora o 3´ splice site (AG) situadas, respectivamente, en los dos nucleótidos iniciales y finales de cada intrón. Una mutación en estas secuencias canónicas (AG/GT) es claramente patogénica. Otras variantes intrónicas y exónicas también pueden alterar el mecanismo de splicing pero su patogenicidad a priori no es tan clara.
La variabilidad clínica entre los pacientes que presentan mutaciones de splicing se ha relacionado con niveles variables del transcrito mutado que pueden venir determinados por dos factores. El primero es si la mutación de splicing altera las secuencias canónicas de splicing (AG/GT) y el segundo es si la alteración del splicing da lugar a una proteína truncada o no truncada. El segundo factor viene definido por si a consecuencia del splicing anómalo se elimina o se inserta en la secuencia codificante un número de nucleótidos múltiplo de 3 o no. En varones afectados de síndrome de Alport ligado al X se han estudio estos dos factores y se ha reportado que el fenotipo es más grave tanto si las mutaciones alteran las secuencias canónicas de splicing [26] como si las mutaciones de splicing generan una proteína truncada [27].
g) Regulación epigenética
La epigenética estudia los cambios en la expresión de los genes que no se pueden atribuir a alteraciones de la secuencia de DNA y que son hereditarios. Hay dos tipos principales de modificaciones epigenéticas: la metilación del DNA y la modificación de las histonas. Las modificaciones epigenéticas actúan como interruptores que encienden y apagan la expresión de un gen (que se traduzca o no a proteína). Cualquier tipo de célula tiene patrones epigenéticos especializados y en caso de enfermedad también cambian los patrones epigenéticos. La epigenética es un campo emergente de la ciencia, por el momento poco estudiado en las ERH.
h) Factores ambientales
El fenotipo está influenciado durante el desarrollo embrionario y durante toda la vida por factores ambientales. Los factores ambientales son muchos y variados e incluyen la dieta, el clima, las drogas, las enfermedades y el estrés, entre otros. Aunque se sabe poco específicamente sobre los factores ambientales y las ERH, es ampliamente conocido, por ejemplo, que una dieta inadecuada puede causar obesidad, diabetes e hipertensión y que estas condiciones agravan la ERH.
7. UTILIDADES CLÍNICAS DEL DIAGNÓSTICO GENÉTICO EN NEFROLOGÍA
Los resultados obtenidos de un diagnóstico genético se caracterizan por ser vitalicios (tienen validez para toda la vida), por tener implicaciones para los miembros de una familia y por influir en la toma de decisiones reproductivas.
Las principales utilidades clínicas del diagnóstico genético son las siguientes:
a) Confirmar un diagnóstico clínico de sospecha es una de las principales utilidades del diagnóstico genético.
b) Reclasificar un diagnóstico clínico p. ej. en un joven con quistes renales y sospecha de PQRAD se puede identificar que está afectado de nefropatía por HNF1B.
c) Filiar una enfermedad renal de etiología desconocida p. ej en pacientes con enfermedad renal crónica diagnosticados en fase avanzada en que se desconoce cuáles eran los signos y síntomas al debut, sobre todo si tienen antecedentes familiares con nefropatía.
d) Identificar un diagnóstico específico dentro de una categoría clínica más amplia p. ej. un paciente con proteinuria y biopsia renal que muestra glomeruloesclerosis segmentaria y focal identificarle una mutación en COL4A4 permite su diagnóstico etiológico preciso de síndrome de Alport autosómico dominante.
e) Diagnóstico presintomático muy útil cuando existe la posibilidad de aplicación de tratamientos preventivos, disminución de riesgos, modificación de hábitos de vida, asesoramiento genético reproductivo, entre otras.
f) Ofrecer un asesoramiento genético preciso que permite informar al paciente sobre su riesgo de transmitir la enfermedad a su descendencia y a sus familiares sobre su riesgo a padecer o transmitir la enfermedad. Permite ofrecer opciones reproductivas como el test genético prenatal o preimplantacional.
g) Estudio de portadores de una enfermedad recesiva tiene importantes implicaciones en las decisiones reproductivas y puede conllevar un futuro diagnóstico prenatal o preimplantacional.
h) Descartar una enfermedad genética en un familiar en estudio p. ej. como potencial donante vivo.
i) Guiar la evaluación de manifestaciones extrarrenales: p. ej. un paciente con mutación en PAX2 debe derivarse al oftalmólogo.
j) Evitar la biopsia renal, p. ej. en pacientes con síndrome de Alport confirmado por diagnóstico genético.
k) Indicar tratamientos específicos o incluir pacientes en ensayos clínicos.
En cuanto a las opciones reproductivas que se hacen factibles si se conoce la causa genética de la ERH se encuentran el diagnóstico genético prenatal y preimplantacional:
El diagnóstico genético prenatal consiste en un análisis genético del feto para determinar si presenta la mutación/es que causan la enfermedad en su familia. Es imprescindible que la familia haya realizado un estudio genético previo al embarazo en el que se haya identificado la mutación/es que causan la enfermedad o los haplotipos ligados a la enfermedad en su familia. Generalmente el análisis genético fetal se realiza a partir de una biopsia de vellosidades coriónicas que se obtiene entre las semanas 10-12 del embarazo. El diagnóstico prenatal normalmente se solicita en enfermedades muy graves con un patrón de herencia autosómico recesivo, como la PQRAR o el síndrome nefrótico congénito. En cambio, para enfermedades autosómicas dominantes con inicio en el adulto la demanda es muy baja. Una cuestión que el especialista debe explicar con claridad a los familiares, es que la solicitud de un estudio prenatal implica una determinación clara de interrupción voluntaria del embarazo en caso de que el feto esté afectado, puesto que se trata de un prueba invasiva con un riesgo de aborto del 0.5-2%.
El test genético preimplantacional (PGT) consiste en la selección de embriones libres de una determinada alteración genética para su transferencia en el útero materno. Es imprescindible que la familia haya realizado un estudio genético previo en el que se haya identificado la mutación/es que causan la enfermedad o los haplotipos ligados a la enfermedad en su familia. Antes de iniciar el proceso la pareja debe superar un estudio de la aptitud (ginecológico y andrológico) para técnicas de reproducción asistida y, por otro lado, debe realizarse un estudio de informatividad que consiste en la adecuación de las técnicas de genética molecular para detectar el defecto genético en unas pocas células o única célula con una fiabilidad que supere el 90%. Superados estos pasos se inicia el procedimiento de fecundación in vitro. El análisis de los embriones se realiza a partir de una sola célula extraída de embriones en estadío de 8 células el tercer día de cultivo embrionario, o bien a partir de 3-6 células extraídas de embriones en estadío de blastocisto el quinto día de cultivo embrionario. A partir del análisis genético realizado a las células embrionarias es posible seleccionar aquellos embriones que no presentan la alteración genética familiar para su transferencia al útero materno. De este modo, la probabilidad de una descendencia sin esta enfermedad será superior al 90-95%. No obstante, dado que los errores técnicos asociados al análisis de una única célula no son menospreciables, la European Society of Human Reproduction and Embriology (ESHRE) recomienda la realización de un diagnóstico prenatal del feto.
Debido a las implicaciones del diagnóstico genético, siempre debe ir acompañado de consejo o asesoramiento genético, tal y como se establece en la Ley de Investigación Biomédica del año 2007 que determina el marco legal del diagnóstico genético.
El asesoramiento genético es un proceso de comunicación mediante el cual un profesional especializado asesora a un paciente con una posible enfermedad genética. Durante este proceso, se ofrece información sobre la enfermedad, el riesgo de padecerla o transmitirla, prevención, tratamientos y opciones reproductivas, con el fin de que pueda tomar sus propias decisiones de forma autónoma.
Hoy en día debemos aprender a seleccionar los pacientes en los que está indicado un estudio genético para tratar de identificar las variantes genéticas causantes de la enfermedad y poder trasladar estos hallazgos hacia una medicina más personalizada. Por otra parte, el estudio genético nos ayuda a mejorar el estudio de la patogenia de una enfermedad, nos brinda información sobre el pronóstico y el manejo clínico de la misma.
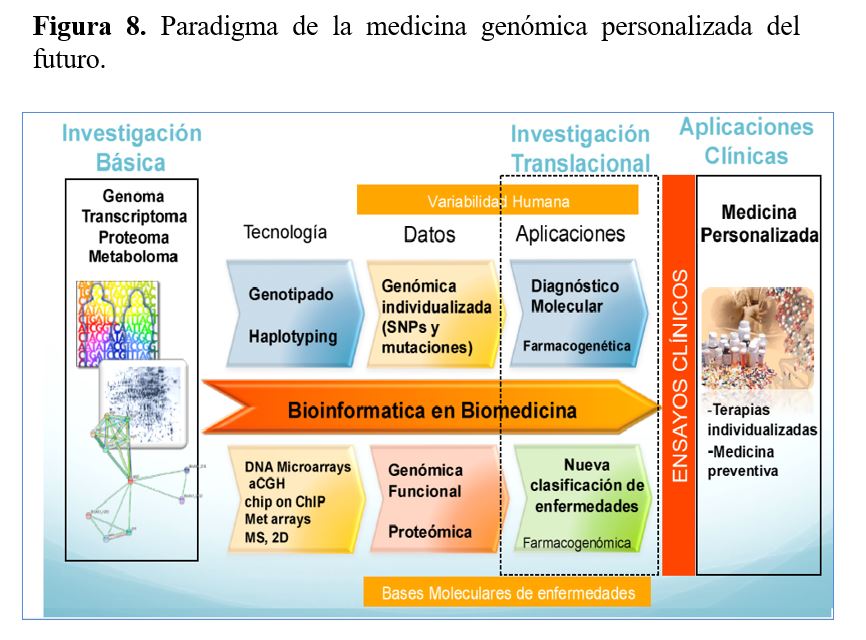
Todo ello nos está acercando a la medicina genómica personalizada cuyo objetivo es la atención individualizada a cada paciente en base al conocimiento de su clínica, su genoma y sus influencias ambientales (Figura 8). En relación con el tratamiento, la farmacogenética tiene el objetivo de ayudar a determinar el fármaco o conjunto de fármacos más indicados para cada paciente concreto y en las dosis adecuadas, en base a sus datos clínicos y genéticos, para mejorar la eficacia y eliminar o disminuir los efectos secundarios de otras terapias más generalistas. Todos estos avances en el conocimiento de las bases moleculares de las enfermedades implican un nuevo paradigma de la medicina en el que los estudios "-ómicos" (genómicos, proteómicos y metabolómicos) tanto de los individuos sanos como de los afectados por distintas enfermedades será analizado junto con su historia clínica y utilizado para predecir las enfermedades antes de que se desarrollen. La medicina genómica predictiva y personalizada tiene el potencial de transformar la asistencia sanitaria a través de la prevención, el diagnóstico precoz y el tratamiento individualizado.
WEBGRAFÍA
ClinVar, http://www.ncbi.nlm.nih.gov/clinvar
GeneReviews; http://www.ncbi.nlm.nih.gov/books/NBK1116
Genome Aggregation Database (gnomAD), http://gnomad.broadinstitute.org
Human Gene Mutation Database, http://www.hgmd.cf.ac.uk/ac/index.php
Human Genome Variation Society (HGVS), http://www.hgvs.org/mutnomen
Leiden Open Variation Database (LOVD); http://databases.lovd.nl/shared/genes
Online Mendelian Inheritance in Man (OMIM), http://www.omim.org
Orphanet, http://www.orpha.net/consor/cgi-bin/index.php
PKD Mayo database, http://pkdb.mayo.edu